Vous pouvez l'attendre :quelque part dans le monde, une nouvelle maladie virale hautement contagieuse et dangereuse se déclare. De quel virus s'agit-il ? Est-ce une nouvelle variante d'Ebola ou du VIH ? Ou le virus Zika ? A quelle vitesse mute-t-il ? D'où vient-il ? Comment est-il transféré ? Y a-t-il aussi des porteurs sans symptômes de maladie ? Quels sont les points de départ prometteurs pour un vaccin ou un médicament ?
Cela aide avec toutes ces questions si vous pouvez cartographier rapidement et complètement la composition génétique du virus. C'est beaucoup plus difficile avec un virus qu'avec une espèce animale ou végétale, car les virus mutent beaucoup plus vite. Souvent, plusieurs souches du virus apparaissent déjà chez un patient au cours de la maladie.
Jasmijn Baaijens – aujourd'hui postdoctorant à la Harvard Medical School de Boston, mais qui a obtenu son doctorat en septembre du Center for Mathematics and Computer Science d'Amsterdam – a développé divers algorithmes pour mieux analyser l'ADN des virus. "J'ai commencé cette recherche parce que ce type d'algorithme m'attirait vraiment", dit-elle. "C'était aussi très d'actualité, car il y avait à l'époque une importante épidémie d'Ebola en Afrique de l'Ouest."
Cartographier l'ADN d'un virus n'a pas été une tâche aisée. En comparaison, la lecture du génome (la séquence complète d'ADN) des humains, des animaux ou des plantes est très claire :l'ADN de toutes les cellules est le même, et les différences – les mutations – entre les individus d'une même espèce sont relativement rares. Un génome de référence est également déjà connu pour l'homme et de nombreuses espèces animales et végétales, dont les individus ne diffèrent que peu.
La lecture d'un génome peut être comparée à l'assemblage d'un puzzle. Vous devez d'abord couper tout l'ADN avec des enzymes en petits morceaux. Ces morceaux lâches qui se chevauchent partiellement - les lits – sont lisibles, mais après cela, vous devez reconstituer tout le puzzle – avec des milliers, voire des millions de pièces. Cela aide énormément s'il existe un génome de référence. Ensuite, il y a une plaque d'échantillon sur le couvercle de la boîte avec des pièces de puzzle, qui diffère tout au plus dans les détails. Sans génome de référence, vous avez une boîte de pièces de puzzle sans plaque d'échantillon. Assembler un tel puzzle s'appelle déterminer un génome "de novo".
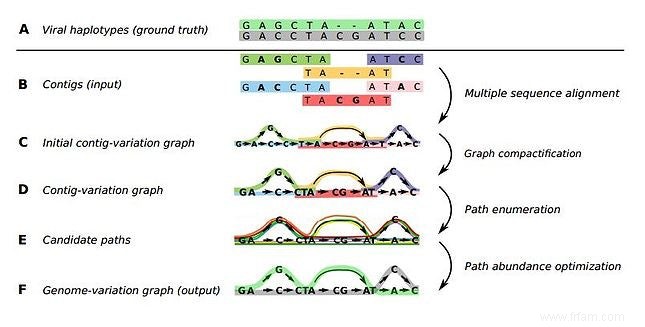
Reconstruire un génome à partir des reads est en fait un problème purement mathématique :vous disposez d'un grand nombre de fragments d'environ mille lettres (le code génétique n'utilise que quatre blocs de construction différents, symbolisés par les lettres C, G, A, T). Certains de ces fragments se chevauchent, car un grand nombre de copies du génome sont coupées dans le tube à essai avec des enzymes - une molécule d'ADN est beaucoup trop petite pour être étudiée de cette manière. En examinant de près les fragments qui se chevauchent, vous pouvez découvrir comment le génome est structuré. Si quelqu'un lise par exemple se termine par ……. GTTAGCT , et un autre commence par AGCT AGC……, cela suggère qu'ils sont adjacents dans le génome intact, de sorte qu'il s'agit d'un fragment …….GTTAGCT AGC……. contient. De cette manière, un ordinateur doté d'un algorithme de recherche efficace peut trouver toutes les lectures, ou autant de lectures. puzzle ensemble en un tout.
En pratique, bien sûr, il est plus indiscipliné. Tout d'abord, tous les séquenceurs font (les machines qui lisent les fragments d'ADN) font parfois des erreurs, il y a donc des "erreurs d'orthographe" dans les lectures siéger. Il est également possible que toutes les copies du génome ne soient pas exactement identiques. C'est certainement une possibilité si vous analysez un échantillon de sang d'un patient qui contient des milliers de particules virales qui ont déjà muté en différentes souches. Supposons que l'ordinateur reconstitue deux longs fragments d'ADN viral de dix mille lettres identiques à l'exception de quatre lettres :est-ce le même fragment avec quatre fautes d'orthographe, ou les deux fragments appartiennent-ils à des souches de virus différentes ?
Dans sa thèse, Baaijens présente divers algorithmes qui utilisent la théorie des graphes pour répondre à ces questions mieux et plus rapidement qu'il n'était possible jusqu'à présent. "Grave" est le terme mathématique pour un réseau de nœuds. Pour déconcerter les lectures . ensemble Baaijens a utilisé un soi-disant graphique de chevauchement (voir image ci-dessous). L'idée d'utiliser des graphes de chevauchement n'est pas nouvelle, mais elle était tombée en désuétude car pour les grands génomes, comme les humains, elle était trop gourmande en calculs pour être d'une utilité pratique. Cependant, elle a pu rendre cette méthode adaptée au génome relativement petit des virus, bien qu'elle ait encore dû développer de nouvelles techniques d'optimisation pour s'assurer que le temps de calcul était maintenu dans des limites.
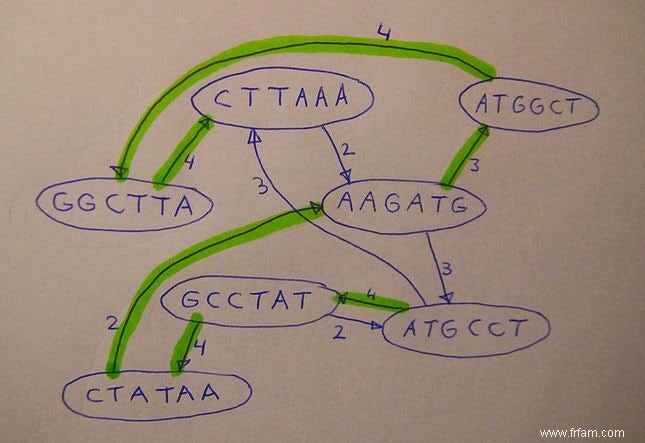
Illustration ci-dessus :Principe du graphe de chevauchement. Les morceaux uniques (les nœuds du graphique) sont reliés par une flèche si la fin d'un morceau chevauche au moins deux lettres avec le début d'un autre morceau. Si vous trouvez un itinéraire complet à travers tous les nœuds, il y a de fortes chances qu'il s'agisse du "génome" d'origine, dans ce cas ATGCCTATAAGATGGCTTAAA.
Le résultat est un algorithme, SAVAGE (Strain Aware VirAl Genome assEmbler ), avec lequel le génome viral dans toutes ses variantes peut être déterminé avec une grande précision, y compris les pourcentages dans lesquels chaque variante se produit. Bien sûr, cet algorithme a ensuite été testé sur différents ensembles de données d'ADN viral connues, et les résultats se sont avérés nettement meilleurs que ceux des algorithmes déjà existants dans ce domaine.
La recherche scientifique se termine trop souvent par des résultats préliminaires et la principale conclusion selon laquelle des recherches supplémentaires sont nécessaires. Pas dans ce cas. Baaijens soutient qu'avec cette thèse, un problème sérieux est essentiellement simplement résolu :la détermination d'un génome viral complet avec toutes ses variantes "de novo" (littéralement :de nouveau, donc sans connaissance préalable, "à partir de zéro").
Néanmoins, bien sûr, d'autres développements sont possibles. Par exemple, supposons qu'un génome contienne un fragment répété qui est plus long que les lectures. Ensuite, vous obtenez un cycle (un anneau fermé de nœuds) dans le graphique. Après tout, le chemin qui décrit le génome complet passe deux fois par le même nœud, à savoir celui avec le fragment répété. Baaijens :"C'est un problème très difficile qui n'est pas encore résolu par notre algorithme."
Le nouvel algorithme peut également être utilisé pour cartographier le génome de cellules tumorales en mutation rapide chez un patient. Dans certains cas, une chimiothérapie ou une immunothérapie peuvent alors être adaptées à cela, afin que la tumeur soit combattue plus efficacement.
Il appartient maintenant aux chercheurs médicaux d'ajouter cet outil à leur arsenal. Baaijens :« Je pense que le logiciel est à un stade où il peut déjà être utilisé par les chercheurs, mais il y a encore beaucoup de points à améliorer. J'espère pouvoir développer davantage le logiciel afin qu'il devienne adapté à une utilisation générale. Au cours de mes recherches doctorales, j'ai consulté occasionnellement des chercheurs en virus qui s'intéressaient particulièrement au VIH, afin de mieux comprendre leurs problèmes et leurs intérêts. Le logiciel peut être téléchargé gratuitement, le code est également open source, donc n'importe qui peut l'adapter et le développer lui-même."
Elle-même va désormais travailler à Boston sur la reconstruction du génome des bactéries :"En fait un problème similaire à celui des virus, mais à une toute autre échelle." Les bactéries mutent également rapidement, mais leur génome est beaucoup plus grand.